本文概述
数据丢失在每个数据科学家的日常工作中都会造成问题。我们应该归咎于他们吗?如果可以, 哪种方法合适?还是可以简单地删除缺少数据点的观测?为了回答这些问题, 人们需要知道缺失数据背后的机制是什么。用统计测试检测它很复杂, 有时只会导致含糊其词。相反, 可视化工具易于使用, 不仅有助于检测缺失的数据机制, 而且有助于深入了解数据质量的其他方面。在本教程中, 将介绍VIM软件包中提供的一组绘图方法, 以显示它们如何帮助人们扎实地掌握数据丢失方式中的哪些模式。
数据机制缺失
数据集不完整可能有多种原因。调查丢失数据的可能原因至关重要, 因为这会影响我们解决问题的方式。例如, 如果担心与工作相关的调查中没有回答, 那么人们可能会期望最富有和最贫穷的受访者不要在问卷中披露其收入, 这意味着缺失的数据点不会在数据集中平均分布。如果是这种情况, 在进行感兴趣的分析之前简单地删除不完整的观测值将产生偏差的结果。另一方面, 例如, 如果由于数据收集设备的故障而导致数据丢失, 则可能是数据集中缺失点的位置纯粹是随机的。
可以根据三种不同的模式来丢失数据。它们通常被称为丢失数据机制。
- 完全随机丢失(MCAR)在MCAR下, 数据集中丢失数据点的位置没有系统的模式:它们完全随机发生。从形式上讲, 这意味着缺少观察的概率既不取决于其他变量的值, 也不取决于其自身的值。在这种情况下, 丢弃不完整的观察结果不会对后续分析的结果造成偏见。示例:温度是由传感器连续测量的, 该传感器收集数据并将其通过Internet发送到数据库。由于未知原因, Internet连接有时会中断。
- 随机缺失(MAR)在MAR之下, 特定观察缺失的概率仍独立于其自身的值, 但是它确实取决于其他变量的值。在这种情况下, 删除不完整的观察值将使样本代表性下降。示例:由于传感器的维护工作而导致的某些夜间时间数据丢失, 而这些维护工作通常在夜间进行。
- 随机丢失(MNAR)在MNAR下, 观察丢失的可能性取决于其自身的未观测值。同样, 在这种情况下, 丢弃不完整的数据会导致分析有偏差。示例:传感器在-20摄氏度下冻结, 并且未测量低于此值的温度。
在实践中, 很难说出这三种缺失数据机制中的哪一种在特定情况下适用。为了看到这一点, 让我们绘制一些MNAR数据。我们将使用之前需要加载的ggplot2和gridExtra软件包。我们还将加载另外两个软件包供以后使用:VIM用于丢失数据的可视分析, 而dplyr用于某些数据预处理。如果你的工作区中仍然缺少它们, 请记住先安装它们。
library("VIM")
library("dplyr")
library("ggplot2")
library("gridExtra")
让我们创建一个具有两个不相关的正态分布变量x和y的数据集, 在其分布的右尾有y的某些缺失值, 这是其最高值之一。请注意, 我们还将保留实际值, 并将其设置为单独变量中的缺失值。
set.seed(2)
mnar_data <- data.frame(x = rnorm(100), y = rnorm(100)) %>%
mutate(y_miss = ifelse(y > 1, y, NA), y = ifelse(is.na(y_miss), y, NA), x_miss = ifelse(is.na(y), x, NA))
现在, 我们将绘制两个x与y的散点图:一个包含我们假装缺失y值的点, 另一个包含仅观察到的数据。
grid.arrange(
# Plot highlighting missing data points
ggplot(mnar_data, aes(x, y)) +
geom_point(size = 4, alpha = 0.6) +
geom_point(aes(x, y_miss), col = "red", size = 4, alpha = 0.6) +
geom_hline(aes(yintercept = y_miss), col = "red", alpha = 0.6, linetype = "dashed") +
ylim(NA, max(mnar_data$y_miss, na.rm = TRUE)) +
ggtitle("Where the data points are missing"), # What data scientist can see
ggplot(mnar_data, aes(x, y)) +
geom_point(size = 4, alpha = 0.6) +
geom_vline(aes(xintercept = x_miss), col = "red", alpha = 0.6, linetype = "dashed") +
ylim(NA, max(mnar_data$y_miss, na.rm = TRUE)) +
ggtitle("What the data scientist can see"), # Arrange the two plots side-by-side
ncol = 2
)

从第一张图可以清楚地看出数据是真正的MNAR, 数据科学家只能看到缺失点的x坐标。从第二个情节可以得出什么结论?数据是MCAR吗?可能是的, 缺失值的位置似乎在x轴上平均分布。还是他们MAR?经过仔细检查, 对于x的负值, y的缺失值似乎比正数要多, 因此也可能是这种情况。最后, 永远不能排除MNAR, 因为要知道数据是否为MNAR, 根据定义, 我们需要观察未观察到的值。
幸运的是, 我们不必全力以赴。数据可视化工具可以为我们提供有关丢失数据中存在哪些模式的一些指导。现在, 我们将基于第五十八套软件包中的传记图像数据集分析一些有助于检测这些模式的图, 其中包含有关传记胶片的一些信息。
数据清理
在继续进行绘图之前, 数据需要进行一些预处理。下面的代码块将完成此工作。 dplyr管道开始处的select调用会提取目标变量, 并将随后的mutate组与种族相关联, 以便在每个组中具有足够的观察值。然后, 调整一些变量的类:对于字符串变量, 我们需要因子, 对于逻辑变量, 我们需要整数。最后, 为了使图表易于阅读, 将变量重命名为较短的名称。
data(biopics, package = "fivethirtyeight")
biopics <- biopics %>%
select(country, year_release, box_office, number_of_subjects, type_of_subject, subject_race, person_of_color, subject_sex) %>%
mutate(subject_race = ifelse(grepl("^Hispanic", subject_race), "Hispanic", subject_race), subject_race = ifelse(grepl("^African", subject_race), "African", subject_race), subject_race = ifelse(grepl("^Middle", subject_race), "Mid Eastern", subject_race), subject_race = ifelse(subject_race %in% c("White", "Asian", "African", "Hispanic", "Mid Eastern", "Multi racial", NA), subject_race, "other")) %>%
mutate(country = as.factor(country), type_of_subject = as.factor(type_of_subject), subject_race = as.factor(subject_race), subject_sex = as.factor(subject_sex), person_of_color = as.integer(person_of_color)) %>%
as.data.frame()
colnames(biopics) <- c("country", "year", "earnings", "sub_num", "sub_type", "sub_race", "non_white", "sub_sex")
我们最终得到一个由八个变量组成的数据集:
- 国家-电影的一个或多个原籍国,
- 发布年份-
- 收益-美国票房的总收益,
- sub_num-电影中精选的主题数,
- sub_type-主题的职业或被认可的原因,
- sub_race-主题竞赛,
- non_white-指示有色人种的虚拟变量,
- sub_sex-主题的性别。
聚合图
要问的第一个基本问题是:缺少哪些变量观察值, 有多少?聚合图是回答这些问题的有用工具。下面的一线便是你所需要的。
aggr(biopics, numbers = TRUE, prop = c(TRUE, FALSE))
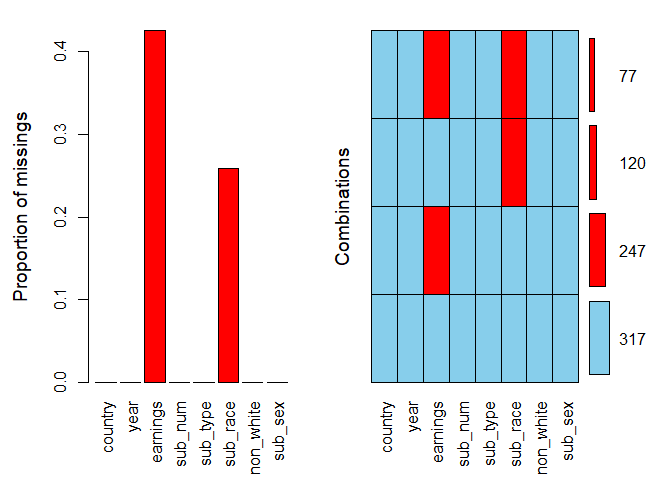
我们指定参数numbers = TRUE在条形图的顶部显示数字。参数prop是逻辑指示是否应使用缺失值和组合的比例而不是总量。对于第一个图, 我们将其设置为TRUE, 对于第二个图, 将其设置为FALSE。
显然, 缺失值仅出现在两个变量中:它们构成收入的40%以上, 约占sub_race的25%。在右侧的组合图中, 网格显示了数据中存在的所有缺失值(红色)和观察值(蓝色)的所有组合。有317个完整的观测值, 在77行中, 两个变量均缺失。
同时查看两个图可能看起来有些混乱, 因此让我们使用以下代码将相同信息强制为一个可视化文件。
aggr(biopics, combined = TRUE, numbers = TRUE)
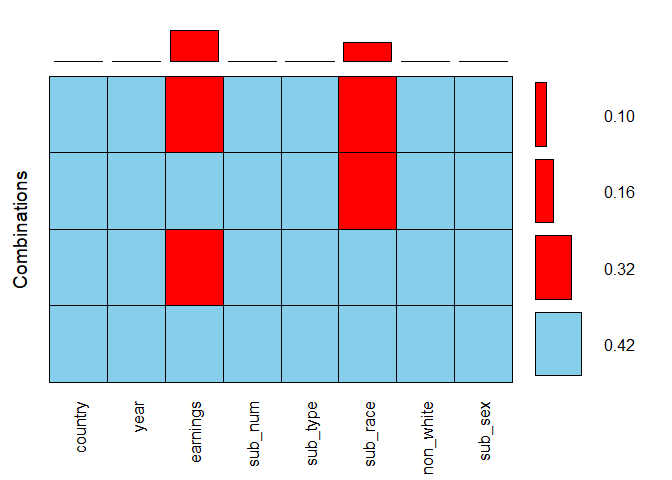
组合参数允许将两个图组合为一个。在此, 网格右侧的水平条显示了相应组合的频率, 而其顶部的垂直条则显示了每个变量中缺失值的比例。通过将所有可能的收入组合的值相加, 即0.32和0.10, 我们可以看到, 该变量中缺失值的总体比例(根据之前的柱状图估计超过40%)实际上为42% 。
自旋图和自旋图
到目前为止, 我们已经对丢失的数据进行了高级概述。现在是时候仔细研究特定变量之间的相互作用了。旋转图和旋转图可以研究一个变量中另一个变量的不同值的缺失值百分比。如果后者是一个数字变量, 那么我们必须处理一个自旋图。当归类时, 此可视化称为自旋图。
两者都可以使用函数spineMiss()生成, 将两列数据框作为输入。第一个指定变量是根据其拆分数据的变量。它被映射到绘图的水平轴上。第二个变量是我们感兴趣的缺少数据模式的变量。为了在实践中看到它, 让我们创建一个旋转绘图。我们首先指定类别变量sub_race, 然后指定收入, 你可以将其读取为:每种sub_race类别的收入中缺失值的百分比是多少?
spineMiss(biopics[, c("sub_race", "earnings")])

sub_race中特定类别的条的相对宽度反映了数据集中该类别的频率:例如, 在绝大多数电影中, 主要主题是白色。在每个条形图中, 显示了收入的缺失比例, 而右侧的阴影条显示了整个数据集的比例。似乎当主要主题是非洲时, 我们最有可能获得完整的收入信息。
通过像下面的代码行中那样切换变量的顺序, 我们可以生成一个自旋图, 以回答以下已解决的问题:sub_race中不同收入值的缺失数据所占的百分比是多少?
spineMiss(biopics[, c("earnings", "sub_race")])

由于收入是一个数字变量, 因此其值被划分为多个bin, 其中bin的宽度与变量本身的分布相对应。这张自拍图显示, 收益严重负向倾斜:只有几部电影获利最高。有趣的是, 对于那些轰动一时的公司来说, 我们很可能会错过主题的竞赛, 这是最重要的收益值最高的红色标线所反映的。
马赛克图
上面讨论的旋转图和旋转图可以研究两个变量之间的相互作用。这个想法以镶嵌图的形式推广到更多变量。此图是图块的集合, 其中每个图块都对应于两个(或多个)变量的类别(用于分类变量)或箱(用于数字变量)的特定组合。在每个图块中, 显示了另一个变量中丢失的数据点的百分比。原则上, 可以为任意数量的变量创建镶嵌图, 但是如果使用太多变量, 它们可能会变得混乱。此外, 如果拆分变量是因子而水平变量又不是太多, 则它们更具可读性。
让我们看一下收入中缺少的部分, 该收入被sub_sex和US_movie分割。后者在下面的代码块中创建, 从逻辑上说明了美国是否参与了电影的制作。我们提供三变量数据框作为mosaicMiss()的第一个参数。参数plotvars设置为具有元素1和2的向量意味着我们要根据该数据帧的前两列拆分数据。将Highlight设置为3意味着我们希望第三列中缺少的变量比例显示在图块内部。
biopics <- biopics %>%
mutate(US_movie = ifelse(grepl("US", country), TRUE, FALSE))
mosaicMiss(biopics[, c("sub_sex", "US_movie", "earnings")], highlight = 3, plotvars = 1:2, miss.labels = FALSE)

同样, 图块的大小对应于数据集中给定组合的出现频率。例如, 最大的右下角图块表示大多数电影都带有男性主题, 并且至少部分是在美国制作的。
对于以男性为主要主题的电影, 非美国电影更可能缺少收入信息。另一方面, 如果主角是女性, 则美国电影的收益价值略有缺失。但是, 这些差异似乎太小而不能视为重大差异。
平行箱线图
另一种可视化类型是并行箱线图。其背后的想法是将数据集分为两个子集:一个仅具有不完整变量的观测值, 而另一个仅具有缺失值。对于这两个子集, 都会生成一个选定数字变量的箱线图。这使你可以检查所选变量的分布是否受拆分变量的缺失值影响。
为了产生一个平行的箱线图, 我们可以使用pbox()函数。我们只传递一个参数:一个两列的数据帧。方框图将在第一列中绘制变量, 而第二列中的变量将用于拆分。在下面的示例中, 我们采用收入的对数, 因为它们高度偏斜, 如果没有这种转换, 则箱形图将类似于一条直线。
biopics <- biopics %>%
mutate(log_earnings = log(earnings))
pbox(biopics[, c("log_earnings", "sub_race")])

左侧的白框显示log_earnings的总体分布, 蓝色和红色分别显示sub_race中观察值和缺失值的子集的分布。框的相对宽度反映了它们所基于的子集的大小:蓝色框较宽意味着在sub_race中观察到的值多于缺失值。除此之外, 这两个盒子看起来彼此相似, 整体看起来也很相似。这表明缺少种族信息不会影响收入分配。
平行坐标图
我们已经研究了单个变量及其相互作用。现在让我们一次分析数据集中的所有变量。允许这样做的可视化是平行坐标图。在该图中, 每个变量被转换为相同的比例并由平行轴表示。在分类变量的情况下, 坐标轴的比例分为一组等距的点, 每个类一个。缺失值放置在垂直轴上方, 在绘图区域之外。绘图上的每一条线对应于数据中的一个观测值, 线的颜色表示所选变量中的缺失数据。
我们可以使用parcoordMiss()函数生成平行坐标图。默认情况下, 它将使用提供的数据框中存在的所有变量作为其第一个参数。将高亮显示在收益上会以不同的颜色标记代表观测值的线, 其中收益缺失值。将alpha混合设置为0.6可使线条稍微透明, 从而增加绘图的可读性。
# Remove variables created for the previous plots
biopics <- biopics %>%
select(- US_movie, - log_earnings)
parcoordMiss(biopics, highlight = 'earnings', alpha = 0.6)

在上图中, 深红色线表示观测值中缺少收益。这些突出显示的观察结果似乎与其余数据不同。特别是, 只有少数几个对应于该国第二高的水平, 否则它包含许多观察结果!事实证明, 这是美国/英国电影(要看到这一点, 只需运行关卡(biopics $ country)并寻找倒数第二个值!)。
此外, 对年轴的快速检查显示出一个咒语, 其高度约占其高度的三分之二, 而暗红色线条的交叉点较少。似乎不久之前, 收入数据更加完整了。这表明国家和年份都可能有助于解释收入中缺失值的分布!
矩阵图
我们将检查的最后一个可视化工具是矩阵图。它通过矩形可视化数据矩阵的所有单元格。观察到的数据以连续的灰黑色配色方案显示(颜色越深, 值越高), 而缺失的值以红色突出显示。优良作法是按不完整变量之一对数据进行排序-这使图形更易于解释。下面的代码块完成了该工作, 并按收入排序。
matrixplot(biopics, sortby = c('earnings'))

该图验证了以前的一些发现:收入值缺失的观测值往往来自很久以前(年份的低值), 并且在国家/地区中得分也很低-这由以下两行中颜色较浅的颜色表示哪些收入是红色的。同样, 当缺少收入时, 看起来有非白色主题的电影也更少了。
结论
综上所述, 似乎传记资料数据集中缺少的数据不是MCAR。自旋图和自旋图证明, 收入和sub_race中缺少数据点的位置可以相互解释。此外, 矩阵图和平行坐标图表明, 国家, 年份和非白人也可能有助于解释收入中缺失值的分布。因此, 删除不完整的观察结果不是一个好主意, 因为这很可能会给我们的推断带来偏差。
最后的想法
你已经走了很长一段路!现在, 你知道缺失的数据机制是什么以及它们之间有何不同, 更重要的是, 如何使用各种可视化工具对其进行推断。做得好!现在, 你已经具备了分析自己不完整数据集的全部条件!
如果你有兴趣了解有关R的更多信息, 请参加srcmini的ggplot2数据可视化(第1部分)课程。
 srcmini
srcmini
评论前必须登录!
注册